2024年3月12日,美国斯坦福大学Michael P. Snyder院士团队(周欣博士和申小涛博士作为共同一作)以封面文章的形式在Cell Host & Microbe发表题为Longitudinal profiling of the microbiome at four body sites reveals core stability and individualized dynamics during health and disease的重磅研究。(查看热心肠日报短科普)

Cell Host & Microbe封面
(2024年4月刊)
该研究作为整合人类微生物组项目(iHMP)的重要组成部分,完成了迄今最完整的纵向人类微生物组计划蓝图。它详细阐述了人体不同部位的微生物组在6年跟踪期内的稳定性,并且利用多组学数据,进行了针对微生物-免疫、微生物-代谢、微生物-临床指征等一系列整合分析。
热心肠日报特别专访第一作者、美国斯坦福大学医学院Michael Snyder院士课题组成员周欣博士,深入解读iHMP研究,分享背后鲜活的科研故事。
专家访谈
Q:祝贺周博士发表重要成果!这项研究是美国整合人类微生物组项目(iHMP)的一部分,可否请您先介绍一下iHMP的具体情况?这项研究在iHMP中的定位是怎样的?
周欣博士:非常荣幸能够有机会借助热心肠研究院的平台交流我们的工作。
2000年前后,人类基因组计划完成以后,全球顶尖测序机构都在寻求能够释放如此大量测序能力的课题,各国先后开展了包括1000人基因组、模式动物基因组测序的一系列工作。
2007年,美国国立卫生院在George Weinstock的建议下,启动了人类微生物组计划(HMP)。第一期计划希望能够通过对人体微生物组进行测序和拼装,找到人类微生物组的“黄金标注”。第一期招募了大约300名年龄在18~40岁的健康志愿者,在18个不同的身体部位对将近1.2万个微生物组样本进行了深度测序。然而事与愿违,并没有找到一个对所有人都适用的、具有普遍性的健康微生物组图谱。
2014年,美国国立卫生研究院希望通过对微生物组的跟踪,比较在特定疾病下的微生物人体动态互作,了解微生物组失调是否参与慢性疾病的发生和发展。当时锚定了3类疾病,分别是炎症性肠病、前驱糖尿病和早产。
与第一期不同的是,第二期HMP计划在微生物组的基础上,要对宿主同时进行深度测序。在人类多组学领域有丰富研究经验,发明并改进了包括RNA-seq、ChIP-Seq、ATAC-Seq的斯坦福大学遗传学系主任Micheal Snyder院士,在2014年加入第二期HMP的工作,这项工作被命名为“整合人类微生物组计划”(Integrative Human Microbiome Project,iHMP)。
两期HMP共耗时10年,耗资2.15亿美元(数据来源:NHGRI),产生近650篇高水平研究论文,被引超过7万次,极大推进了人类对共生微生物的认知,开启了延续至今的一个全新研究领域。
Q:请您讲讲这项研究背后的心路历程,以及与您的博士导师、iHMP研究的牵头人George Weinstock院士的故事?
周欣博士:这个工作得益于我的博士导师George Weinstock院士、博后导师Michael Snyder院士的常年合作和对我的悉心指导,我想趁这个机会讲讲这项工作背后的故事。
George引领我走上微生物组研究之路
我和George相识是在微生物学的课堂上,当时他在介绍微生物组的工作,恰逢人类微生物组计划第一批数据发表,介绍完这批数据的产生和用途后,他故作神秘地笑着说:“大家知道吗,其实我们研究微生物组和它的功能,解释了很多以前无法解释的神奇现象。比如中医里,药材的产地导致药性的不同,很大程度上是由产地环境微生物组差异所导致的发酵过程差异引起的。” 这个理论让我感觉大为新奇,课间就去找他讨论,他说: “欣,不如你来我的课题组做一篇博士论文,我保证你能对微生物组有新的认识!我对中医的了解,大多是好友赵立平教授(现新泽西大学应用微生物系主任)告诉我的。这真神奇,我们应该研究它!”

图1 George Weinstock院士
2014年,我加入了George课题组,攻读博士学位。正值美国国立卫生院启动人类微生物组计划第二期的数据采集,着重研究肠道微生物对糖尿病、炎症性肠病和早产的影响。
进入课题组后,我开始学习基础的分析和实验技能,因我有一些实验基础,上手很快,但是生信分析总是很难提高。当时实验室的师兄师姐们,手把手地教我做分析。我经常坐在陈磊师兄(现上海交通大学教授)身边,看他编译代码。
课题组的最初几年,我们刚刚开始样本收集工作,并没有开展大规模的测序和分析,我总是和George讨论我应该做点什么,并且表达对文章和毕业有些焦虑。某天中午吃饭和George闲聊,他说:“你应该做你觉得好玩的课题,不用太把文章和毕业放在心上。科研必须由兴趣驱动!”
渐渐地,我开始探索一些“好玩”的方向。我们观察了很多有趣的微生物组,比如病毒感染对肠道细菌的影响(Bartley & Zhou,et al. Fron Immu,2017),母鼠如何把肠道菌通过母乳传递给子代(Zhou,et al. Gastroenterology,2017),以及他当年上课提到的,不同产地的中药材的环境微生物组对比(Xia & Zhou,et al.BMC Microbiology,2019)等等。
通过这样的简单观察,我很快进入了角色,并且逐渐参与了生物统计学工具的开发(Yang & Johnson & Zhou. Sci Rep,2019)。另外还有诸多未发表的观察,比如对FXR敲除小鼠(肠道屏障损伤模型)的肠道微生物组进行分析,竟然机缘巧合得到了该模型的权威Sridhar Mani教授的关注。Mani教授一眼就认出了我当年的研究,还用邮件发来当年我提交给George的测序数据和研究报告,并提出指导我继续他和George在8年前未完成的合作。
当这篇Cell Host & Microbe文章的数据采集工作还没有完成的时候,我在George课题组已经开始着手进行一些简单的分析。我们观察到,在胰岛素抵抗的患者,几乎很难检测到Th17细胞相关的细胞因子,他们的肠道微生物多样性远低于正常人,并且体内缺乏梭菌纲的细菌。而在小鼠体内,梭菌纲恰恰是促进Th17细胞发育的。
我们觉得这样的巧合背后一定有相关的生物学原理,于是我提出了一个假设:在胰岛素抵抗患者,肠道缺乏梭菌的患者(约占糖尿病患者的6%)可以用补充梭菌来介导Th17细胞的发育,从而让这类细胞发挥修复肠道表皮的功能,并且修复肠道上皮可能是逆转胰岛素抵抗的关键驱动因素。
实际上,这类干预手段在小鼠已经取得了令人震惊的成功。于是我们提出了两个观点:第一,部分2型糖尿病患者携带因Th17细胞失活导致的免疫缺陷;第二,针对这类的患者,细菌移植疗法将会是一个很有前景的干预手段。
美国糖尿病协会杂志刊发了我们的研究(Zhou & Johnson. Diabetes,2020),也让我开始真正对精准医学,尤其是大数据驱动下的个性化医疗产生了兴趣。 这类的数据驱动,后来又让我发表了一系列基于共同细胞/分子表型(Yu & Zhou. Circulation Research,2022)和并发症组(Rockson & Zhou,et al. Clinical and Translational Medicine,2022)相关的疾病与精准医疗研究。
在Diabetes文章发表之前,George被确诊食道癌,起因是他在参加北京的肠道大会时晕倒,回到美国就进行了全面体检。噩耗震惊了整个课题组,所有人都为George捏了一把冷汗。我和几位师兄师姐也商量好,这个关键时刻我们谁也不离开课题组,一起陪George到手术结束。

图2 团队焦急等待George手术康复后回归实验室
(摄于2018年8月15日,美国康涅狄格州杰克逊实验室)
手术一波三折,确诊后发现肺部的癌细胞转移了,在肺叶切除的手术中又感染了耐药金黄葡萄球菌。所幸手术还算成功,细菌感染也控制住了。George回到实验室的第一句话就是:感染我的那个耐药菌,当年的基因组还是我完成的!我们听闻都笑做一团,阴霾也很快散去。
主动出击,去斯坦福做博后
很快到了博士第五年,当时我主要的工作还没有发表,正逢George的博导David Botstein院士(现Calico首席科学家)来实验室参观,George就鼓励我去介绍自己的工作,但是我上前支支吾吾。后来George叫我去他办公室,严肃地对我说:“你的工作相当优秀,你应该敢于展示自己的工作,不要因为工作没有发表就畏缩。这会让David觉得你对自己的研究缺乏自信。”
我觉得自己怯场实属不该,然而刚刚才给David留下了这个印象,总不好意思马上联系他。于是就鼓起勇气向刚刚来组里访问过的Michael Snyder院士发了一封邮件,介绍了我们尚未发表的工作,并且询问了博士后的职位。两小时后竟然收到了Mike的邮件,信里只有3句话:“非常欢迎你来做我的博士后!我了解你的工作,所以不需要面试。你什么时候来斯坦福报到?”我的博士答辩结束后,George很高兴地说斯坦福的遗传学系是他当年做博士后的地方,我去那里做博士后,一定要多拍一些照片给他。
告别了George, 我带着成堆的未知,准备去旧金山湾区开始博士后工作。无巧不成书,他当年博士后工作的实验室,和我将要工作的实验室仅一墙之隔。2023年我回康州拜访他,给他看了很多实验室照片,他都还清晰地记得当年每个座位上坐着谁,以及他们(我的各路师叔们)在斯坦福的轶事。临别时,George对我说:“斯坦福的经历将是你最值得回忆的经历。所以到了湾区,你千万别一头扎进实验室,要多出去走走看看,多想想你的科研方向,你想做什么!当年我刚到斯坦福,就足足打了半年的高尔夫,直到湾区的雨季来临,我才开始我的课题。”这和我们复旦“自由而无用”的民间校训颇有异曲同工之妙。
后来到Mike课题组里报道,在周文郁师姐(现Adaptive Biotechnology公司总监)的帮助下,我很快熟悉了环境。师姐当时完成了最后一批数据的测序和整理工作,再加上George课题组的测序数据,构成了这篇Cell Host & Microbe文章的完整数据集。当时去和Mike汇报数据时,他说:“你应该创造一个教科书式文章,让我们对这批数据的解读成为以后微生物组的必读文章之一。”为了不辜负他的期待,我阅读了几乎所有的相关论文,以至于最后这篇文章引用了将近300篇文献,几乎和我的博士论文持平。
Cell Host & Microbe文章的研究心路
我当时想到的第一个出发点,就是菌群的长期稳定性。这个稳定性在动物模型中很难观察,因为动物实验环境相对固定。其他课题组对人类的肠道微生物稳定性做过一些基础的工作,但是多个部位菌群的稳定性,目前没有很好的定量研究,也缺乏和宿主联系的解读。
基于这个原因,我们引入了“核心微生物”的概念,即分析哪些微生物在6年的时间内总是稳定出现。我们发现在胰岛素抵抗患者,肠道核心微生物群的数量是显著下降的,缺失的部分主要是负责代谢产生短链脂肪酸的菌群;皮肤核心微生物群的数量反而上升,增加的几个核心微生物都是机会致病菌(图3)。而这种差异在正常人感染类似流感的病毒时也可以短暂观察到。
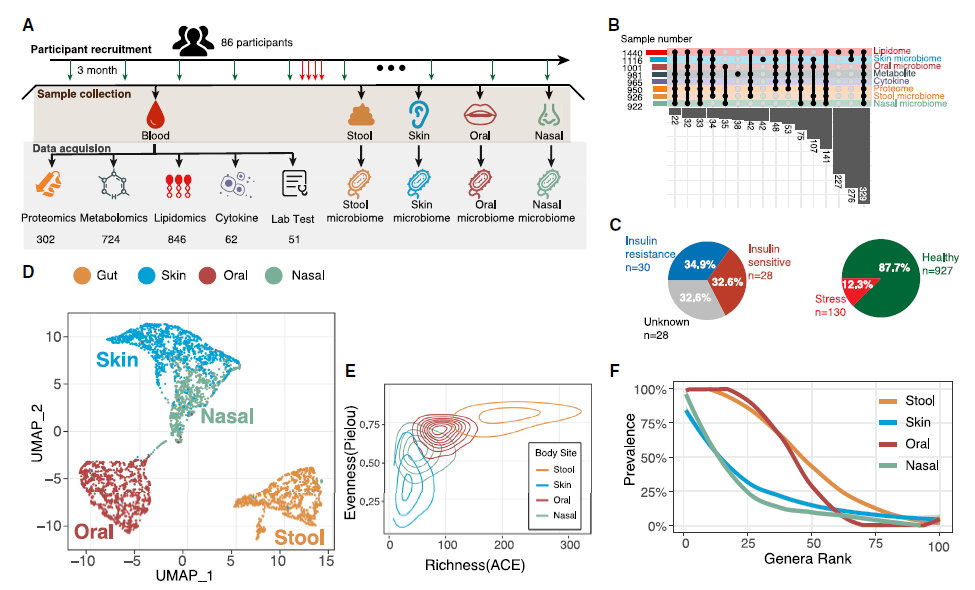
图3 4个身体部位微生物组的纵向分析
(引自 DOI: 10.1016/j.chom.2024.02.012)
我们在这个认知的基础上谋求更系统的观察,想探索是否每个人的菌群都是不一样的,而这又是另一个仅能在人类队列研究的指标。这个工作的思路来源于傅静远教授团队此前在Cell发表的肠道微生物指纹图谱的工作,我们计划实现微生物个体差异的定量分析,并将微生物的个体差异和稳定性联系起来。
所以我设计了一个DMI(Degree of Microbial Individuality)的定量统计指标,来定义每个微生物属的个性化程度。
我们还意外地发现,拟杆菌门细菌更容易进化出个性化强的种类(图4)。而且,和我们猜想的一样,个性化程度越强的细菌,其稳定性也越强(图5)。也就是说,人体有一套非常强的“选择系统”,强力控制到底什么样的菌群可以定植在宿主体内或体表。

图4 对于不同的属和身体部位,微生物组的个性化有显著差异
(引自 DOI: 10.1016/j.chom.2024.02.012)

图5 与个性化和应激事件相关的微生物组时间稳定性
(引自 DOI: 10.1016/j.chom.2024.02.012)
整合多组学分析,破解“选择系统”
为了在分子层面上破解这个“选择系统”,我们利用宿主的高精度多组学测量数据,从免疫、代谢两个角度对宿主和微生物的互作进行了分析。
我的师兄Jethro(现牛津大学教授)和Daniel (现俄亥俄州立大学教授)开发了一套基于贝叶斯统计马尔科夫链蒙特卡洛方法的模型,可以精确计算出免疫系统和微生物的互作。
基于这个模型和代谢组、脂质组等多组学的关联分析,我们发现在这个选择系统中,核心微生物的选择大概率是依靠营养供给(代谢),非核心微生物的选择则更多依靠免疫反应,由此构成了我们的免疫-微生物互作(图6)和代谢-微生物互作(图7)。
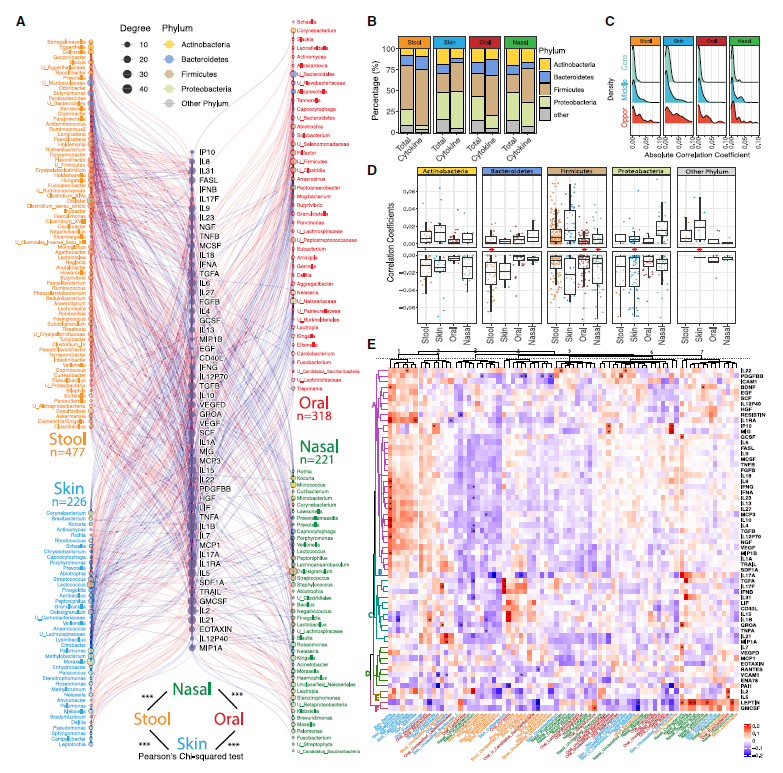
图6 循环细胞因子与微生物组的系统联系
(引自 DOI: 10.1016/j.chom.2024.02.012)
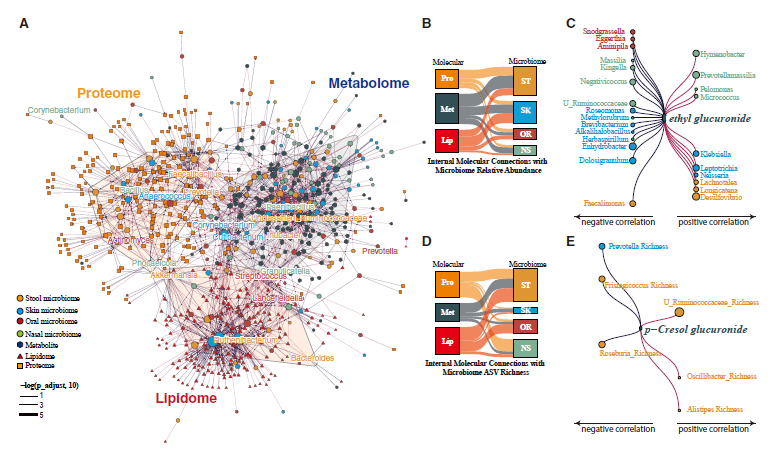
图7 血浆代谢物、脂质、蛋白质组学和微生物组随时间的相互作用
(引自 DOI: 10.1016/j.chom.2024.02.012)
了解到这个强力“选择系统”的存在,我们想进一步更深入地探究微生物组在胰岛素抵抗的发生和发展中的作用。当时是2021年,正好因果推断众望所归,并斩获了当年的诺贝尔经济学奖。
我发现,其实因果推断可以完美地从这个“选择系统”过滤出微生物组为因、临床指标为果,而免疫、代谢为中介诱因的通路。
我们就借用了因果推断的“中介分析”来研究这个问题。果不其然,印证了我们在Diabetes文章中提出的假设,胰岛素抵抗患者广泛缺失由细胞因子介导的肠道微生物对免疫系统的调控,而患者的口腔、鼻腔微生物可能更多地促进炎症产生(图8)。
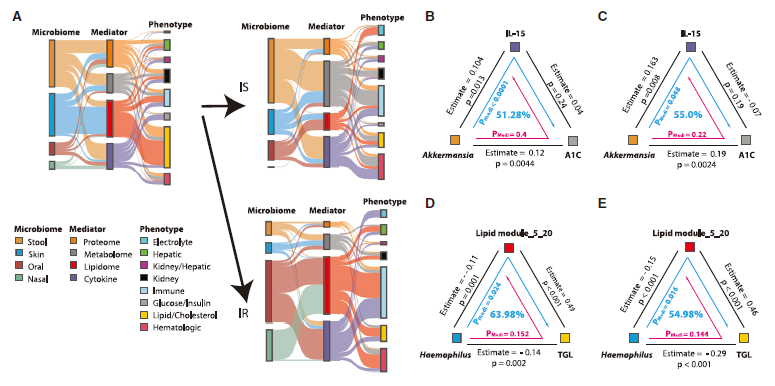
图8 因果推断解码由内部分子和细胞因子介导的微生物组驱动的表型动态
(引自 DOI: 10.1016/j.chom.2024.02.012)
未来的研究征程
我们的工作不仅仅能将多组学大数据驱动下的个性化医疗推动到定量的层面,并且针对2型糖尿病和胰岛素抵抗的菌群干预规划了一个分子蓝图。
人类微生物计划的样本和大数据目前还保存在斯坦福大学,我计划今年5月留任斯坦福大学医学院遗传学系担任研究科学家,未来将带领团队对样本库中的单菌株进行分离、鉴定、研究,通过大数据分析、结合人工智能来指导我们利用菌株进行精准的个性化临床干预,并在菌群与免疫互作,尤其衰老、抗肿瘤等领域与其他团队展开广泛合作。

图9 2018年George实验室合影
不幸的是,在这项研究工作被Cell Host & Microbe接收前的几个月,George与病魔斗争了近7年,虽然跑赢了5年生存率,但还是于2023年11月25日不幸离世。我于2023年6月和George的会面,不想竟成永别,对当时没有合影颇为遗憾。这篇文章是他生前的最后工作,也为他的伟大科学家生涯画上了圆满的句号。
谨以此文,纪念人类微生物组计划的发起人、领导者,微生物组的奠基人之一,科学先驱,我的博士导师George Weinstock院士。
专家简介

周欣
博士,美国斯坦福大学医学院Michael Snyder院士课题组成员,致力于研究大数据、多组学,尤其是肠道微生物组与人体的互作图谱,并利用人工智能工具发掘其与人类健康的关系。
本科毕业于复旦大学,博士受教于肠道微生物领域奠基人之一、人类微生物组计划发起人George Weinstock院士,博士后加入斯坦福大学医学院Michael Snyder院士课题组。
过去3年,陆续获得美国国立衰老研究院青年科学家基金、比尔及梅琳达·盖茨基金会斯坦福新药孵化定向基金、斯坦福-以色列RAMBAM国际合作项目等多个基金资助。
担任包括Molecular Psychiatry、Biofilms and Microbiomes等中科院一区杂志评审专家,以及Frontiers in Bacteriology、Infectious Microbes & Diseases等杂志编委。
迄今在包括Nature、Cell Host & Microbe、Circulation Research等杂志发表学术论文28篇,被引用超过1000次。